Monosomies et trisomies : de nouvelles données pour établir les chances de réussite à la pose des embryons
Les anomalies chromosomiques, comme les monosomies (perte d’un chromosome) et les trisomies (présence d’un chromosome supplémentaire), ainsi que d’autres anomalies comme les polyploïdies (présence de plusieurs copies complètes du génome) ou les haploïdies (présence d’un seul jeu de chromosomes au lieu de deux), peuvent compromettre le développement embryonnaire et la viabilité des animaux. Pour les détecter, nous avons choisi d’exploiter des données jusqu’ici indisponibles pour la recherche et donc inutilisées provenant des puces de génotypage. Ces outils, déjà largement utilisés pour analyser le patrimoine génétique des bovins, permettent également de mesurer la quantité d’ADN associée à des fragments chromosomiques spécifiques.
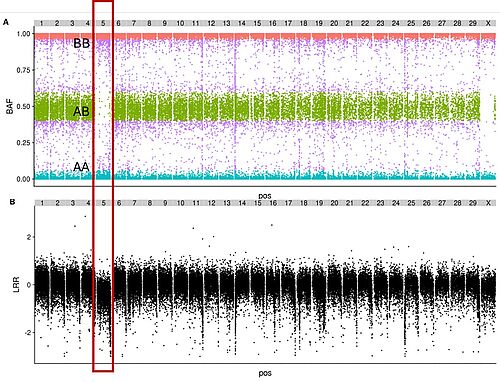
Comprendre les données de fluorescence
Pour identifier les anomalies, nous analysons les informations issues de la fluorescence détectée par les puces à ADN utilisées dans le cadre de la sélection génomique à l’aide de deux indicateurs clés :
- La BAF (B Allele Frequency) qui mesure la proportion de l’allèle B par rapport à l’allèle A (Figure A)
- La LRR (Log R Ratio) qui mesure l’intensité de fluorescence et reflète donc la quantité d’ADN présente pour chaque fragment chromosomique (Figure B)
Une méthode fiable pour détecter les anomalies
Grâce à un premier ensemble de données comprenant 192 embryons produits in vitro, nous avons développé une méthode robuste pour classer chaque chromosome de chaque embryon comme étant normal, monosomique ou trisomique. Ce travail a nécessité de relever plusieurs défis, notamment l’adaptation de la méthode à la qualité souvent moindre des données de génotypage embryonnaire par rapport à celles des adultes. Après avoir testé différentes approches, nous avons sélectionné une méthode capable de détecter les anomalies même à partir du seuil minimal de qualité attendu pour un génotypage de l’embryon.
Ces premiers résultats sont prometteurs : la méthode sera bientôt validée sur de nouveaux jeux de données, ce qui permettra d’affiner encore les outils et les analyses.
Des applications prometteuses pour la sélection …
L’analyse des données complémentaires issues du génotypage ouvre des perspectives intéressantes pour améliorer la sélection génomique. Un des objectifs du projet est d’intégrer ces informations dans les systèmes actuels, permettant à terme d’appliquer la méthode de détection d’aneuploïdie chez l’embryon, mais aussi de développer des tests pour d’autres anomalies chromosomiques moins délétères mais pouvant avoir un impact sur la carrière et la productivité des animaux (voir les exemples de la thèse Fertili’Gest).
… et la compréhension des anomalies chromosomiques
Les embryons produits in vitro de race Holstein et issus de la technologie OPU-FIV (Ponction Ovocytaire suivie de Fécondation In Vitro) seront également analysés grâce aux données de fluorescence. De plus, chaque embryon OPU_FIV sera suivi en morphocinétique (système Primo-Vision® présent à la station ELIANCE de Nouzilly) afint de suivre en temps réel leur développement. En analysant des événements cellulaires précis (comme la durée des mitoses ou la survenue d’anomalies de développement) et en les corrélant aux anomalies chromosomiques observées (monosomie, trisomie…), nous espérons identifier très précocement des embryons aneuploïdes et ainsi mieux comprendre les mécanismes à l’œuvre.
Pour en savoir plus
Le projet AnEmBoV est un projet financé par APIS-GENE avec un partenariat entre l’UMT eBis pour les analyses génétiques, la station expérimentale Eliance de Nouzilly et l’équipe BREED (INRAE) pour la production et l’analyse morphocinétique des embryons.
Contacts : clementine.escouflaire(at)eliance.fr ; daniel.lebourhis(at)eliance.fr