
Valoriser les races bovines locales grâce au séquençage du génome entier
Conserver la diversité génétique des races à petits effectifs…
Jusque dans les années 60, le cheptel français était très diversifié avec plus de 40 races avec des effectifs importants. La sélection moderne a favorisé la spécialisation et la diffusion des quelques races les plus productives. Si une quinzaine d’autres races adaptées à un contexte régional ont pu garder une population de taille modérée, de nombreuses populations bovines locales ont vu leur taille de population tomber à un niveau très bas et n’ont évité l’extinction que par la mise en place de programmes de conservation. Aujourd’hui, il existe de multiples enjeux (environnementaux, économiques et culturels) autour de la conservation des races locales à petits effectifs.
…pour la pérennité du cheptel bovin Français
Le maintien de la diversité génétique intra-race, mais aussi entre races, est indispensable pour faciliter l'adaptation rapide aux changements potentiels d’orientation de la sélection des animaux domestiques. Dans l’avenir, la grande diversité des races bovines françaises sera un atout majeur pour adapter la sélection aux enjeux associés au changement climatique, avec une importance grandissante des caractères associés à l’adaptation à des milieux difficiles et à la résilience.
Le séquençage des génomes offre de nouvelles opportunités
Avec l’avènement de nouvelles technologies de séquençage de génomes entiers, la séquence totale de l’ADN, désormais économiquement plus accessible, est devenue un outil de choix pour évaluer les différences génomiques entre les races. Ces nouvelles technologies représentent ainsi un outil incontournable pour caractériser les populations d’élevage et pour mieux comprendre les mécanismes moléculaires qui permettent à ces races de s’adapter à leur milieu de production.
Ainsi, le séquençage d’individus appartenant à cinq races françaises à petits effectifs (Armoricaine, Froment du Léon, Maraichine, Saosnoise et Lourdaise, Figure 1) a permis de contextualiser les micro-variations de leur génome par rapport à d’autres races bovines françaises majeures et de mettre en évidence des régions génomiques abritant des gènes liés à des caractères de production et/ou d’adaptation spécifiques de ces races (cf. exemple du gène BCO2, plus loin).

Figure 1: Les 5 races françaises à petits effectifs étudiées sont (a) la race Saosnoise, originaire du Nord de la Sarthe, se caractérise par son grand format et sa rusticité, elle est issue de croisements de taureaux Shorthorn et Normands avec des vaches de race Mancelle (bovins peuplant le Maine, l’Anjou et la Touraine au 18e siècle) ; (b) la race Maraichine, race mixte qu’on retrouve dans la branche fauve du rameau brun avec les races Aubrac, Nantaise, Parthenaise et Tarentaise, est issue d'une ancienne population appelée "poitevine" ou "vendéenne", originaire de l'ouest de la France (Marais côtiers atlantiques) et sélectionnée pour le labour, elle est connus pour sa rusticité et son adaptation aux milieux humides ; (c) la race Armoricaine, créée au 19ème siècle à partir de croisements entre les races Froment du Léon, Pie Rouge de Carhaix (disparue) et la race anglaise Durham, se caractérise par un dépôt de gras assez important (viande très persillée, de qualité) et une rusticité qui lui permet d’être bien adaptée au froid ; (d) la race Froment du Léon est une race laitière créée dans le Pays de Léon (nord de la Bretagne) qui produit un lait riche en matière grasse et en bêta-carotène, ce qui donne au beurre une couleur jaune très prononcée ; (e) la race Lourdaise, originaire des Hautes Pyrénées, appartient au grand ensemble des races blondes du Sud-Ouest (Limousine, Blonde d'Aquitaine, Bazadaise et Béarnaise), elle a été utilisée dans les années 60 pour créer la race Blonde d’Aquitaine et bien que considérée comme une race mixte, la Lourdaise est une des races bovines les plus laitières du Sud-Ouest.
Nous avons utilisé les données de séquence du génome entier de 195 animaux issus de 21 races bovines : les cinq races françaises à petits effectifs ainsi que 14 autres races en majorité françaises à des fins de comparaison. L’objectif était de faire une caractérisation génomique détaillée de ces races. Les 14 races ont été choisies sur la base de leur proximité génétique supposée avec les cinq races Françaises objets de l’étude (selon la littérature). Cette étude conduite en collaboration avec l’université de Aarhus (Danemark) dans le cadre d’Eurogenomics, intégrait également deux races Danoises (Dansk Holstein et la Jutland).
Grâce à ces données, nous avons caractérisé l’ensemble des variations du génome (SNP pour Single Nucleotide Polymorphism). Après un contrôle qualité des données et l’élimination des SNP fortement liés, 569 704 SNP ont été utilisés pour identifier la structure génomique des races de l’étude.
Caractériser la structure génomique de races
Une analyse en composantes principales (ACP) a permis de grouper ensemble les populations génétiquement semblables (Figure 2). Les résultats de l’ACP sont confirmés par un autre type d’analyse visant à caractériser la structure génétique ancestrale des individus (analyse de mélange génétique). La figure 3 montre ainsi que la race Armoricaine est génétiquement proche de la Shorthorn et que la Saosnoise partage une composante ancestrale majeure avec la Rouge des Prés (anciennement appelée Maine-Anjou). Elle confirme également la proximité génétique observée entre la Lourdaise et la Parthenaise d’un côté et les trois races du Sud de la France de l’autre (Limousine, Aubrac et Blonde d’Aquitaine).
Figure 2: Analyse en composantes principales faite sur les 21 races de l’étude en utilisant 569 704 marqueurs génétiques de type SNP. Les deux premiers axes PC1 et PC2 expliquant respectivement 2.22 % et 1.92 % de la variabilité génétique totale sont représentés sur la figure. L’Armoricaine est placée à proximité de la Shorthorn. La Saosnoise, la Rouge des Près et l’Angus forment un groupe. Ces deux dernières races sont proches des trois races du sud de la France : Limousine (LIM), Aubrac (AUB) et Blonde d’Aquitaine (BAQ).

Figure 3: Analyse de mélange génétique (structure génétique ancestrale) de 195 animaux issus des 21 races de l’étude. Les individus sont regroupés par population. Chaque individu est représenté par une barre verticale. Chaque couleur correspond à un groupe (ascendance) génétique différent. Les races sont séparées par des lignes verticales épaisses noires. SHORN : Shorthorn ; ARMO : Armoricaine ; ANG : Angus ; RDP : Rouge des prés ; SAOS : Saosnoise ; AUB : Aubrac ; BAQ : Blonde d’Aquitaine ; LIM : Limousin ; FROM : Froment du Léon ; LOURD : Lourdaise ; PAR : Parthenaise ; MAR : Maraichine ; NOR : Normande ; GALO : Galloway ; DEXT : Dexter ; SCO : ScottishHigland ; RDC : Rouge Flamande ; SDM : Dansk Holstein ; JUTL : Jutland ; HOL : Holstein ; MRY : Pie rouge des plaines.
Détecter des spécificités génomiques des races à petits effectifs
Plusieurs régions chromosomiques sous sélection ont été identifiées pour la race Froment de Léon, en comparant statistiquement le génome de cette race à celui des autres races. L’une d’entre elles contient le gène BCO2, qui code pour une enzyme connue comme étant un régulateur clé du métabolisme du β-carotène (Figure 4). L’étude a permis d’identifier d’autres gènes candidats dans les cinq races à petits effectifs, liés à des caractères de production, mais aussi à l’immunité et au métabolisme.
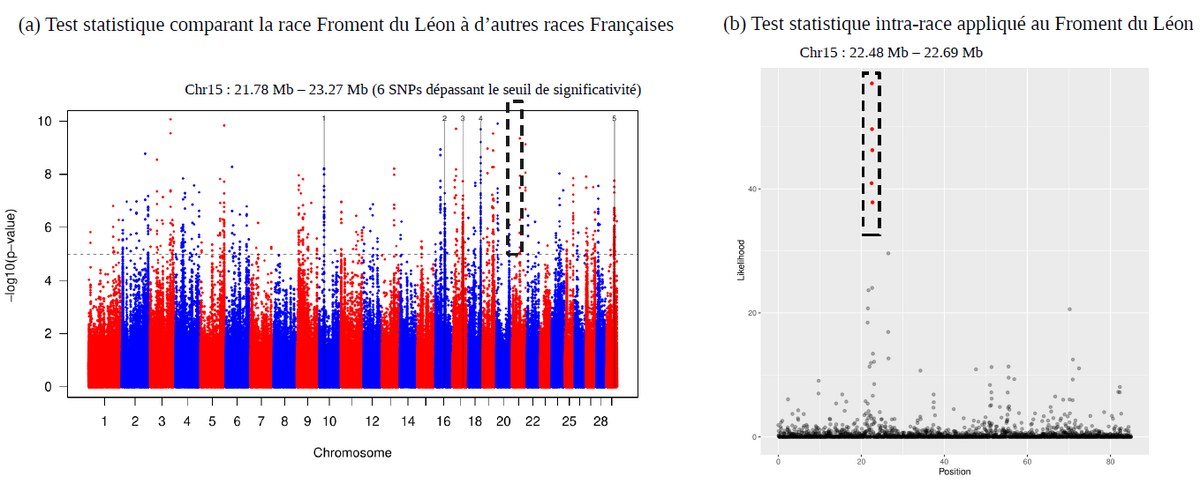
Figure 4: Tests statistiques permettant d’identifier notamment la Bêta-Carotène Oxygenase 2 (BCO2), situé sur le chromosome 15 dans la région 22.51 – 21.58 Mb, comme gène candidat pour la couleur jaune prononcée du lait produit par la race Froment du Léon. (a) Test statistique comparant la Froment du Léon aux trois races du sud de la France (Aubrac, Blonde d’Aquitaine et Limousin). De nombreuses régions, en plus de la région du chromosome 15 contenant le gène BCO2, sont fortement significatives (définies par des SNP qui dépassent le seuil de significativité statistique représenté par la ligne horizontale en pointillés). (b) Test statistique intra-race appliqué sur la Froment du Léon. Les SNP les plus hautement significatifs du chromosome 15 (points en couleur rouge) encadrent la position du gène BCO2.
Une approche pour mieux valoriser les spécificités de chaque race
Cette étude pionnière démontre l’intérêt des études comparatives des génomes de races bovines sélectionnées sous différents environnements et l’importance d’une caractérisation complète et spécifique de chaque race. Dans l’avenir, l’approche pourra être étendue à d’autres races françaises à petits effectifs afin de mieux caractériser les spécificités génomiques de chacune d’elles. Ainsi, les stratégies d'amélioration génétique pourront tenir compte des spécificités de chaque race.
