
EVAGENOC : Vers la mise en place d’évaluation génomique pour les animaux croisés.
Un intérêt croissant des éleveurs pour le croisement

En France, le croisement laitier représente un peu moins de 2% des inséminations artificielles (IA) dans les races laitières mais connait une progression rapide. L’intérêt des éleveurs pour les animaux croisés est lié à leur robustesse, leur rusticité et leurs capacités d’adaptation supérieures (à des changements de système notamment) à celles des animaux de race pure. Le croisement compense les défauts d’une race (faible fertilité, problèmes de santé, format) ou combine les avantages de plusieurs races (haute production et taux intéressants, par exemple) tout en conservant de bonnes performances de production. Des travaux de simulations et des études technico-économiques montrent que la robustesse et la longévité des vaches Holstein croisées est meilleure que celle des vaches de race pure. Le progrès obtenu dès la première génération issue de croisement, résulte de la complémentarité entre les races impliquées dans un croisement et de l’effet d’hétérosis. Enfin, le croisement permet un contrôle efficace des taux de consanguinité et l’augmentation de la diversité génétique au sein d’un troupeau, ce qui en renforce encore la résilience.
Adapter les outils de sélection aux animaux croisés
Les éleveurs d’animaux croisés ne bénéficient pas encore des index génomiques qui permettraient de sélectionner les reproductrices croisées. Une méthodologie d’indexation génomique a donc été développée pour les animaux croisés dans le cadre des projets EVAGENOC (en France) et GENTORE (à l’international). Elle est adaptée aux différents types de croisement et tenir compte des différentes races impliquées pour le croisement rotatif comme pour le croisement terminal. Les travaux portaient sur trois grandes étapes :
- L’imputation des génotypages des animaux croisés,
- La détermination de l’origine raciale des allèles
- La prise en compte de l’origine raciale des allèles dans l’évaluation
Par ailleurs, le génotypage d’un grand nombre d’animaux croisés et de leurs ascendants de race pure était nécessaire pour tester les méthodes ainsi que pour la future mise en place de ces évaluations en routine.
L’imputation, un prérequis nécessaire à toute indexation
L’imputation de génotypages est un prérequis nécessaire et obligatoire avant toute évaluation génomique. Elle a un double objectif :
- Disposer d’informations génomique dans une densité suffisante pour l’ensemble des animaux disposant d’un génotypage (même ceux génotypés sur des puces basse densité).
- Attribuer une origine paternelle et maternelle pour chacun des allèles portés par un individu. Cette étape est appelée « phasage » et elle est importante pour une imputation de bonne qualité. Dans le cas du croisement, elle a un intérêt supplémentaire : la détermination de l’origine raciale des allèles dépend de cette information.
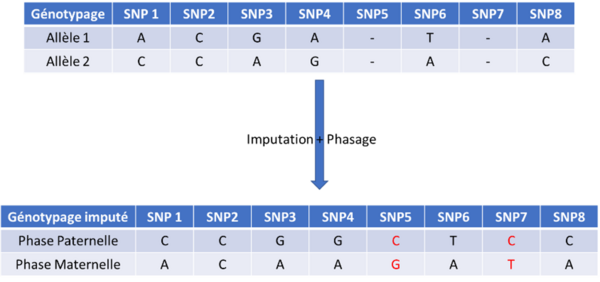
Figure 1 : Principe de l’imputation et du phasage pour un génotypage donné. Avant imputation, le génotype contenait une faible de densité de marqueurs attribués à l’allèle 1 ou l’allèle 2. Suite à l’imputation, les marqueurs manquants ont été complétés par méthodes statistiques. Au cours du phasage chaque variation des marqueurs a été attribuée à une origine paternelle ou maternelle.
Pour les animaux de race pure, l’imputation est réalisée intra-race. Cependant, les animaux croisés sont issus de différentes races et sont apparentés à des animaux de races pures. La méthode d’imputation des animaux croisés intègre donc les génotypages des animaux de races pures et des animaux croisés. L’ensemble des informations disponibles sont ainsi prises en compte pour une imputation de la meilleure qualité possible.
L’effet d’un marqueur génétique varie en fonction de la race

Les évaluations génomiques des animaux de races pures actuellement réalisées en France ont montré que pour un même caractère évalué, l’effet d’un marqueur donné peut varier en fonction de la race. Les animaux croisés étant issus de plusieurs races pures, il est important de tenir compte de la race pure ayant « fourni » les allèles. C’est ce que l’on appelle la détermination de l’origine des allèles ou « chromosome painting ». Cette méthode se base sur la comparaison des fréquences d’haplotypes entre la population des animaux croisés et celles des animaux de races pures.
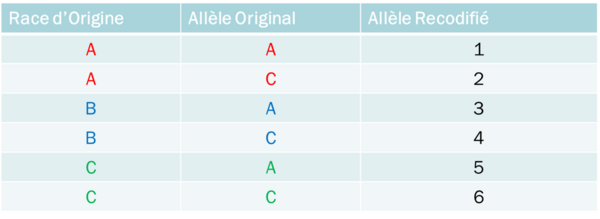
Figure 2 : Détermination de l’origine raciale des allèles et recodification associée des marqueurs d’un génotypage d’un animal croisé (Exemple d’un animal croisé qui porte l’allèle C. Si cet allèle C provient de la race rouge, alors il sera codé « 2 », s’il vient de la race bleue, il sera codé « 2 » et s’il vient de la race verte, il sera codé « 6 »)
Une évaluation génomique adaptée au croisement
Le modèle d’évaluation génomique SNP BLUP a été adapté afin de valoriser les informations issues du chromosome painting. Cette nouvelle version a été implémentée dans un logiciel développé par INRAE et appelée CrossSG. Cette méthode permet d’obtenir pour un marqueur donné autant d’effets pour le caractère évalué que de races pures dont sont issus les animaux croisés étudiés.
6400 animaux croisés génotypés
Les partenaires professionnels impliqués dans EVAGENOC ont été sollicités afin de collecter le matériel biologique nécessaire au génotypage d’animaux croisés. Environ 6400 génotypages d’animaux croisés d’intérêt ont ainsi été obtenus et seront valorisés dans les futures évaluations génomiques.
Une première évaluation test satisfaisante
En partenariat avec le projet GENTORE, 4 380 femelles croisées avec performances, issues des populations Rouge Danoise, Holstein et Montbéliardes ont été évaluées en utilisant la méthode décrite précédemment.
Dans cette population, 94% des allèles portés par les femelles croisés ont eu une origine raciale attribuée : 48% étaient issus de la race Holstein, 34% de la Rouge Danoise et 14% de la Montbéliarde.
Les évaluations génomiques réalisées sur cette population pour les caractères de production ont fourni des précisions très encourageantes pour les animaux candidats (supérieures à 0,30).
Des évaluations génomiques en routine d’ici deux ans
A l’issu du projet EVAGENOC, les méthodes pour l’imputation des génotypages des animaux croisés, pour la détermination de l’origine raciales des allèles ainsi que pour l’évaluation génomique ont été développées et testées avec satisfaction.
La suite de ces travaux consiste à mettre ces éléments à disposition du centre d’évaluation génétique GenEval. Des évaluations d’animaux croisés en routine pourraient ainsi être mises en production à l’horizon des deux prochaines années.






Une évaluation génomique adaptée au croisement
Le modèle d’évaluation génomique SNP BLUP a été adapté afin de valoriser les informations issues du chromosome painting. Cette nouvelle version a été implémentée dans un logiciel développé par INRAE et appelée CrossSG. Cette méthode permet d’obtenir, pour un marqueur donné, autant d’effets pour le caractère évalué que de races pures dont sont issus les animaux croisés étudiés.